2. 临沂市人民医院检验科, 临沂 276000;
3. 济宁医学院生物科学学院, 日照 276826
2. Clinical Laboratory, Linyi People's Hospital, Linyi 276000, China;
3. School of Biology, Jining Medical University, Rizhao 276826, China
乳腺癌是严重威胁女性生命健康的一种疾病,2021年美国女性癌症发病首位为乳腺癌,年发病人数约为28.15万,占女性癌症新发病例的30%;女性癌症死因乳腺癌排第二位,年死亡人数约为4.36万,占女性癌症死亡病例的15%[1]。因此,寻找能够诊断乳腺癌的肿瘤标志物迫在眉睫。目前,三模体家族45(Tripartite Motif Family 45,TRIM45)在乳腺癌中研究较少,通过GDC TCGA Breast Cancer (BRCA)数据库筛选发现TRIM45表达显著上调。TRIM45,一种具有RING-finger结构域的E3泛素连接酶,是TRIM家族的一员,在细胞增殖、分化、凋亡和肿瘤发生发展等多种细胞功能中发挥重要作用[2-6]。TRIM45可通过抑制AP-1和ELK-1转录活性从而负调控MAPK信号传导途径[2];同时,TRIM45是一种新型的RACK1相互作用蛋白,能够抑制RACK1/PKC复合物的形成而下调MAPK信号转导[3];此外,TRIM45可以负调节NF-κB通路并抑制癌细胞增殖[4];并且TRIM45与p53相互作用,通过促进p53的K63连接的多聚泛素化,能够稳定p53,从而抑制了p53的泛素化降解[5]。研究表明TRIM45是一种重要的抗肿瘤分子。因此,本研究主要以单基因TRIM45为研究对象,通过GDC TCGA Breast Cancer(BRCA)数据挖掘,利用生物信息学方法分析TRIM45在乳腺癌中的表达、临床病理相关性及生存预后分析,使用GSEA(Gene Set Enrichment Analysis)基因富集分析TRIM45参与乳腺癌调控的信号通路,揭示其调控乳腺癌发生发展的可能分子机制。
1 材料与方法 1.1 乳腺癌基因芯片表达数据及临床数据的收集和处理乳腺癌患者的临床资料和RNA-Seq数据来自GDC TCGA Breast Cancer(BRCA)(https://xenabrowser.net)。其中,下载1104例乳腺癌组织样本和113例癌旁样本及配对样本(乳腺癌临床样本113例、癌旁样本113例)的临床信息和RNA-Seq数据,所有样本的RNA-Seq数据均已在R软件(V.3.6.0)中使用BioConductor包进行处理和归一化。
1.2 TRIM45在乳腺癌中的表达差异及其与临床病理特征的关系采用R软件(V.3.6.0)对GDC TCGA Breast Cancer(BRCA)数据进行处理,使用edgeR包对RNA-Seq数据作差异分析,计算TRIM45在乳腺癌组织(1104例)和癌旁组织(113例)、乳腺癌组织和与其相匹配的癌旁组织(113例)的差异表达。使用UALCAN (http://ualcan.path.uab.edu)[7]分析TRIM45在乳腺癌中的表达及其与临床病理类型的关系。
1.3 乳腺癌组织TRIM45表达水平与乳腺癌预后的关系按TRIM45在乳腺癌临床样本中的表达均值(FPKM平均值为2.0341)进行分组,低于均值为低表达组,高于均值为高表达组。分析TRIM45表达水平与Overall Survival的关系。
1.4 基因集富集分析使用KEGG基因集[c2.cp.kegg.v7.5.1.symbols.gmt]进行GSEA富集分析,评估TRIM45可能参与乳腺癌调控的信号通路。按TRIM45在乳腺癌临床样本中的表达中位数(FPKM中位数为1.9665),将乳腺癌临床样本分为高、低两组。在分析过程中,TRIM45的表达被认为是高、低两组表型,根据NES(标准化富集分数)、NOM p-val(标准化显著性水平)和FDR q-val(矫正多重假设检验)对每种表型的富集途径进行分类,NES绝对值>1.0,NOM p-val<0.01,FDR q-val<0.05确认为有意义的基因集合。
1.5 统计学方法所有统计分析均使用R(V.3.6.0)软件。两组独立样本数据的比较使用Student′s t-test检验,多组数据间的比较使用秩和检验。采用卡方检验分析TRIM45表达与临床病理特征的关系,使用Kaplan-Meier法作生存分析。以P<0.05为差异有统计学意义。两基因间相关性分析使用Spearman相关性分析,P<0.05并且r绝对值>0.3为差异有统计学意义。
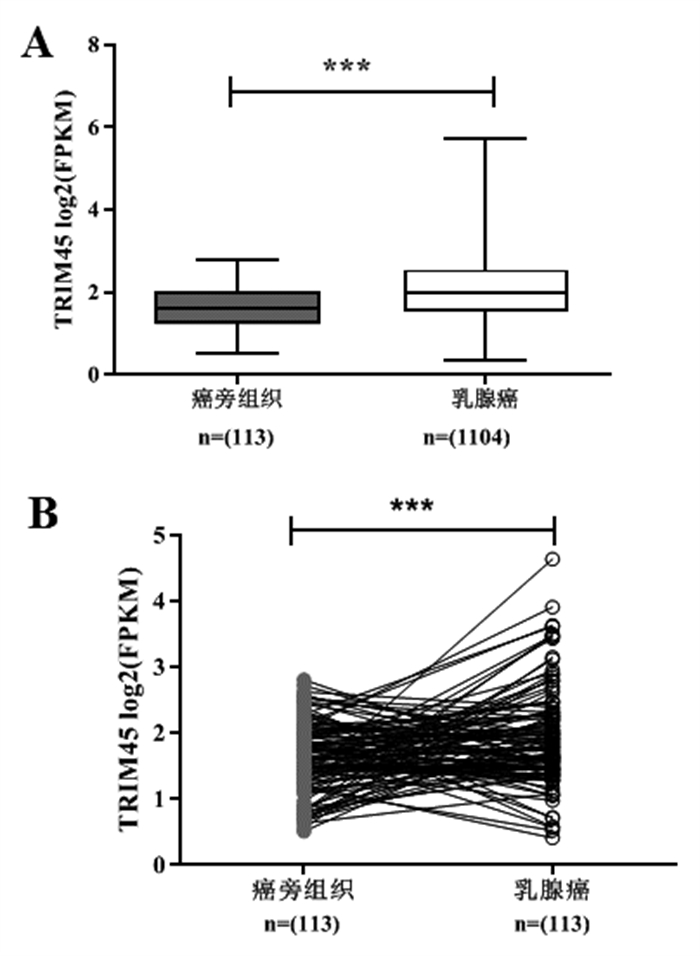
2 结果 2.1 乳腺癌和正常乳腺组织TRIM45差异表达基于TCGA数据分析,在非配对乳腺癌样本与癌旁样本中的差异表达分析显示,TRIM45在乳腺癌中的表达高于癌旁组织(P<0.001)(图 1A)。在配对乳腺癌样本与癌旁样本中的差异表达分析显示,TRIM45在乳腺癌中的表达高于癌旁组织(P<0.001)(图 1B)。
|
图 1 TRIM45在乳腺癌中的表达分析 注:A.TRIM45在乳腺癌组织与癌旁组织中的表达分析;B.TRIM45在乳腺癌组织及其匹配的癌旁组织中的表达分析。***P<0.001。 |
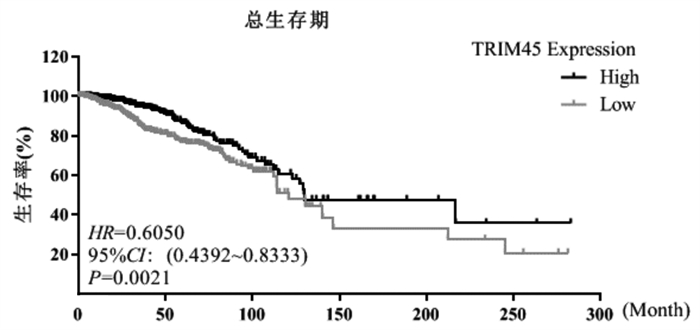
TRIM45高表达的患者总生存期优于TRIM45低表达的患者(P=0.0021)。见图 2。
|
图 2 TRIM45在乳腺癌中的总生存期分析 |
通过对1104例乳腺癌临床数据进行筛选,保留TCGA数据集中包含临床参数和生存资料的病例,最后得到含有完整临床病理参数和生存资料的病例825例,其中TRIM45低表达422例,TRIM45高表达403例。
TRIM45 mRNA表达水平与乳腺癌患者分子分型、T分期、TNM分期以及病理类型存在显著关联(P<0.05)。见表 1。
| 表 1 TRIM45表达与乳腺癌临床病理关联性分析 |
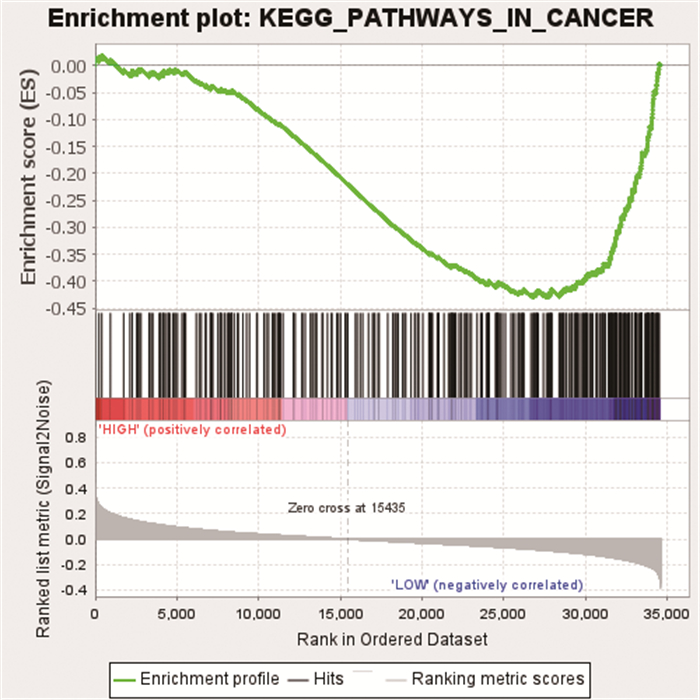
GSEA富集分析发现TRIM45在KEGG_PATHWAYS_IN_CANCER信号通路中发挥重要作用(NES=-1.81,NOM p-val=0.002,FDR q-val=0.023),同时KEGG_PATHWAYS_IN_CANCER信号通路在乳腺癌中发挥重要作用。见图 3。
|
图 3 GSEA信号通路富集分析 |
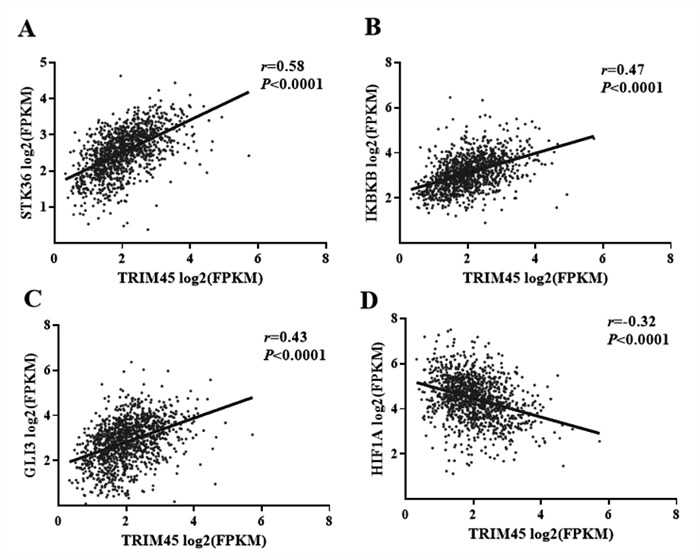
通过相关性分析可以发现TRIM45与KEGG_PATHWAYS_IN_CANCER信号通路中多种基因存在相关关系,其中STK36、IKBKB、GLI3与TRIM45具有正相关关系(图 4A~4C),HIF1A与TRIM45具有负相关关系(图 4D)。
|
图 4 TRIM45表达与KEGG_PATHWAYS_IN_CANCER信号通路共表达基因筛选 注:A.TRIM45与STK36的相关性分析;B.TRIM45与IKBKB的相关性分析;C.TRIM45与GLI3的相关性分析;D.TRIM45与HIF1A的相关性分析 |
TRIM45是一种具有RING-finger结构域的E3泛素连接酶,在细胞增殖、分化、凋亡和肿瘤发生发展等中发挥重要作用[2-6]。本研究通过挖掘TCGA数据明确TRIM45在乳腺癌中表达上调。通过乳腺癌临床病理相关性分析,发现TRIM45在乳腺癌中的表达与T分期、TNM分期、分子分型及病理类型相关。通过生存分析显示,TRIM45高表达乳腺癌患者的总生存期高于TRIM45低表达者,以上生物信息学数据分析结果显示TRIM45有望成为乳腺癌抑癌基因,且TRIM45高表达能显著提高乳腺癌患者的生存率。
为进一步研究TRIM45在乳腺癌中的功能及调控机制,基于TCGA数据利用GSEA软件进行KEGG基因集富集分析。GSEA富集分析发现TRIM45在KEGG_PATHWAYS_IN_CANCER信号通路中发挥重要作用。通过相关性分析可以发现TRIM45与KEGG_PATHWAYS_IN_CANCER信号通路中STK36、IKBKB、GLI3具有正相关关系,与HIF1A具有负相关关系。
STK36是丝氨酸/苏氨酸激酶家族的成员,是GLI锌指转录因子的正调节因子,在Hedgehog信号通路中起关键作用[8-9]。GLI3编码的蛋白质属于GLI家族成员,是一种DNA结合转录因子,并且是Sonic Hedgehog(Shh) 信号传导的介质[8]。Hedgehog信号通路在调控细胞的增殖、分化、干细胞调控、肿瘤的发生中起着关键性的作用[10]。IKBKB编码的蛋白能够磷酸化NF-κB复合物中的抑制剂,导致抑制剂解离并激活NF-κB,进一步活化NF-κB信号通路[11-12]。HIF1A编码缺氧诱导因子-1(HIF-1)的α亚基,而HIF-1是由α和β亚基组成的异二聚体[13-14]。HIF-1作为细胞和系统对缺氧的稳态反应的主要调节因子,参与调控能量代谢、血管生成、细胞凋亡等基因转录[13-15]。
综上所述,通过挖掘TCGA数据库,应用生物信息学技术推测TRIM45可能作为潜在的诊断乳腺癌和预测其预后的肿瘤标记物,其内在的调控机制可能是通过调控STK36、IKBKB、GLI3和HIF1A影响乳腺癌细胞的增殖、侵袭和转移,尚需要进行一系列的实验来验证本文的预测结果,为乳腺癌的早期诊断和疗效评估提供新的靶点。
利益冲突:所有作者均申明不存在利益冲突。
| [1] |
Siegel RL, Miller KD, Fuchs HE, et al. Cancer statistics, 2021[J]. CA Cancer J Clin, 2021, 71(1): 7-33. DOI:10.3322/caac.21654 |
| [2] |
Wang Y, Li Y, Qi X, et al. TRIM45, a novel human RBCC/TRIM protein, inhibits transcriptional activities of ElK-1 and AP-1[J]. Biochem Biophys Res Commun, 2004, 323(1): 9-16. DOI:10.1016/j.bbrc.2004.08.048 |
| [3] |
Sato T, Takahashi H, Hatakeyama S, et al. The TRIM-FLMN protein TRIM45 directly interacts with RACK1 and negatively regulates PKC-mediated signaling pathway[J]. Oncogene, 2015, 34(10): 1280-1291. DOI:10.1038/onc.2014.68 |
| [4] |
Shibata M, Sato T, Nukiwa R, et al. TRIM45 negatively regulates NF-κB-mediated transcription and suppresses cell proliferation[J]. Biochem Biophys Res Commun, 2012, 423(1): 104-109. DOI:10.1016/j.bbrc.2012.05.090 |
| [5] |
Zhang J, Zhang C, Cui J, et al. TRIM45 functions as a tumor suppressor in the brain via its E3 ligase activity by stabilizing p53 through K63-linked ubiquitination[J]. Cell Death Dis, 2017, 8(5): e2831. DOI:10.1038/cddis.2017.149 |
| [6] |
Peng X, Wen Y, Zha L, et al. TRIM45 suppresses the development of non-small cell lung cancer[J]. Curr Mol Med, 2020, 20(4): 299-306. DOI:10.2174/1566524019666191017143833 |
| [7] |
Chandrashekar DS, Bashel B, Balasubramanya S, et al. UALCAN: a portal for facilitating tumor subgroup gene expression and survival analyses[J]. Neoplasia, 2017, 19(8): 649-658. DOI:10.1016/j.neo.2017.05.002 |
| [8] |
Murone M, Luoh SM, Stone D, et al. Gli regulation by the opposing activities of fused and suppressor of fused[J]. Nat Cell Biol, 2000, 2(5): 310-312. DOI:10.1038/35010610 |
| [9] |
Maloveryan A, Finta C, Osterlund T, et al. A possible role of mouse Fused (STK36) in Hedgehog signaling and Gli transcription factor regulation[J]. J Cell Commun Signal, 2007, 1(3-4): 165-173. DOI:10.1007/s12079-007-0014-y |
| [10] |
Patel TN, Dhanyamraju PK. Role of aberrant Sonic hedgehog signaling pathway in cancers and developmental anomalies[J]. J Biomed Res, 2021, 36(1): 1-9. DOI:10.7555/JBR.35.20210139 |
| [11] |
Schmid JA, Birbach A. IkappaB kinase beta (IKKbeta/IKK2/IKBKB)——a key molecule in signaling to the transcription factor NF-kappaB[J]. Cytokine Growth Factor Rev, 2008, 19(2): 157-165. DOI:10.1016/j.cytogfr.2008.01.006 |
| [12] |
Sughra K, Birbach A, de Martin R, et al. Interaction of the TNFR-receptor associated factor TRAF1 with I-kappa B kinase-2 and TRAF2 indicates a regulatory function for NF-kappa B signaling[J]. PLoS One, 2010, 5(9): e12683. DOI:10.1371/journal.pone.0012683 |
| [13] |
Masson N, Willam C, Maxwell PH, et al. Independent function of two destruction domains in hypoxia-inducible factor-alpha chains activated by prolyl hydroxylation[J]. EMBO J, 2001, 20(18): 5197-5206. DOI:10.1093/emboj/20.18.5197 |
| [14] |
Kim EJ, Yoo YG, Yang WK, et al. Transcriptional activation of HIF-1 by RORalpha and its role in hypoxia signaling[J]. Arterioscler Thromb Vasc Biol, 2008, 28(10): 1796-1802. DOI:10.1161/ATVBAHA.108.171546 |
| [15] |
Xu C, Liu X, Zha H, et al. A pathogen-derived effector modulates host glucose metabolism by arginine GlcNAcylation of HIF-1α protein[J]. PLoS Pathog, 2018, 14(8): e1007259. DOI:10.1371/journal.ppat.1007259 |



