柯萨奇病毒B3(CVB3)是单正链RNA病毒,属于肠道病毒,小RNA病毒科,基因组序列全长约7.4kb,有开放读码框架一个。一旦感染本病毒,即会在宿主体内进行大量复制,造成溶细胞性感染,可导致宿主很快出现大量的细胞损伤死亡。CVB3可引发多种疾病,如肝炎、干燥综合征、胰腺炎、病毒性心肌炎[1-2]、扩张型心肌病[3]、无菌性脑膜炎等疾病[4]。目前应用的各种防治方法均未起到满意效果,需要进一步探究更加安全的、效果更好的防治策略。
RNA干扰(RNA interference, RNAi)近年来在抗病毒研究领域展现了巨大的潜力。siRNA对很多种病毒的抑制效果均比较理想[5-7],针对siRNA抑制CVB3复制的研究目前也有很多团队在进行,有研究证实VP1区[8]、2A区[9]、3C区[10]、3D区[11]均可以设计出有效的siRNA序列,Kim等[12]已证实2C区的CRE部位是设计siRNA序列的有效靶点。
本文将设计的siRNA序列转染入HeLa细胞,筛选抑制效果好的siRNA序列,同时探究2C区CRE以外的基因是否可以设计CVB3特异性较高的siRNA序列。
1 材料与方法 1.1 材料 1.1.1 病毒和细胞HeLa细胞由中国农业科学院哈尔滨兽医研究所细胞中心提供。CVB3 H3株是用包含CVB3基因全长的感染性克隆PMSK-1质粒[13](登录号U57056)转染入细胞后获得[14],该病毒是由哈尔滨医科大学的钟照华教授馈赠,将其在HeLa细胞中活化,滴定该病毒滴度为108TCID50/ml。
1.1.2 主要试剂和仪器EntransterTM-R转染试剂购自英格恩生物公司;荧光定量PCR试剂盒Premix Ex Taq TM(DRR039)由宝生物工程有限公司销售提供;LEICA-HC荧光显微镜购自Leica公司。
1.2 方法 1.2.1 病毒TCID50滴定在96孔的细胞培养板中接种HeLa细胞,密度为5×104个细胞/孔,将细胞培养12h后,进行病毒50%组织培养感染剂量(50% Tissue culture infective dose, TCID50)的滴定。稀释病毒,其浓度依次为1×101、1×102、1×103、1×104、1×105、1×106、1×107、1×108、1×109、1×1010,各取100μL分别加入96孔板中,每个稀释度进行了8个重复,第11列和12列为对照,每天在显微镜下观察细胞的病变(cytopathic effect, CPE),记录结果,直至CPE的终点出现。该过程要保证设置的对照组始终保持正常的形态和特征。用Reed-Muench法计算本病毒的TCID50/ml。
1.2.2 siRNA的设计、合成和标记CVB3基因序列在GenBank中已登录,其登录号为U57056,按照Elbashir等[15]及Reynolds[16]的设计原则,在CVB3的3D、3C、2C、2A、VP3、VP2和VP1区设计了siRNA序列共计11条,分别命名为siRNA1-11。增设了用FAM标记的无关siRNA序列设置为阴性对照组siRNA-NC,阳性对照组siRNA-PC选取往年发已表验证的抑制效率较高的siRNA序列[10]。13条siRNA制备合成时3′端均设计了dTdT悬突(序列见表 1)。合成的siRNA序列是干粉状,将其融入DEPC水中,稀释至终浓度为20μM,存于-20℃备用。
| 表 1 siRNA序列和对应靶区域 |
将FAM标记的阴性对照序列siRNA-NC稀释浓度分别为200nM、、150nM和100nM,EntransterTM-R(μL):siRNA(μg)设为4 :2和5 :2,HeLa细胞接到细胞培养板中用不含抗生素的DMEM营养液进行培养,待细胞达到40%左右的融合度时进行siRNA转染,转染siRNA 12h后通过荧光显微镜及流式细胞术技术观察筛选最适宜的转染siRNA的条件。
1.2.4 转染siRNA后感染CVB3将浓度为150nM的siRNA和Entranster TM-R按照2 :5的比例进行混合后转染入HeLa细胞。本次实验共分16组,siRNA-1、siRNA-2、siRNA-3、siRNA-4、siRNA-5、siRNA-6、siRNA-7、siRNA-8、siRNA-9、siRNA-10、siRNA-11组分别转染相应的siRNA序列,siRNA-PC为转染阳性对照组,siRNA-NC为转染阴性对照组,mock为仅加转染试剂Entranster TM-R组,virus only组为HeLa细胞未转染任何物质组。于转染后12h在以上15组HeLa细胞上分别感染0.01MOI的CVB3,sham infected是正常细胞对照组,未转染siRNA和未感染病毒。于病毒感染HeLa细胞36h后收获目的细胞和病毒,检测siRNA对病毒复制的抑制效果。
1.2.5 MTT法检测抑制效率收获16个观察组的目的细胞,分别加入MTT液培养4h,弃去培养液上清分别加入二甲基亚砜,在酶联免疫检测仪A490nm处测量各孔的吸光值。sham infected是正常细胞对照组,将其细胞的活率设定为100%,分别计算各组细胞的存活率。
1.2.6 Real-time RT-PCR检测抑制效率收获目的细胞和病毒采用实时荧光定量RT-PCR进行检测,选取β-actin为内参,将siRNA-NC处理组设置为calibrate定为1,通过相对定量方法,检测设计的11条siRNA序列对CVB3复制的抑制情况。
1.2.7 Western blot检测VP1蛋白表达变化收获目的细胞,处理后样品按20μl/孔上样,分别用β-actin单克隆抗体(1 :5000)和VP1蛋白特异性的多克隆抗体(1 :1000)37℃孵育1h,洗膜后分别加入辣根(HRP)标记的山羊抗鼠IgG二抗和山羊抗兔IgG二抗,孵育处理后,扫膜仪扫膜。
1.2.8 毒价滴定检测病毒抑制率HeLa细胞转染13条siRNA序列12h后感染CVB3,感染病毒36h后收获HeLa细胞,滴定病毒液上清TCID50值。
1.3 统计学方法采用SPSS22.0统计学软件进行数据分析,定量资料用x±s表示,组间比较采用ANOVA,P < 0.05为差异有统计学意义。
2 结果 2.1 转染率的测定在倒置荧光显微镜下观察不同浓度下siRNA-NC的转染率,发现siRNA染率在浓度为200nM和150nM时较高。流式细胞检测结果显示当siRNA浓度为150nM,EntransterTM-R(μL):siRNA(μg)=5 :2时,siRNA转染的效率最好,确定此时为最佳转染条件(图 1)。
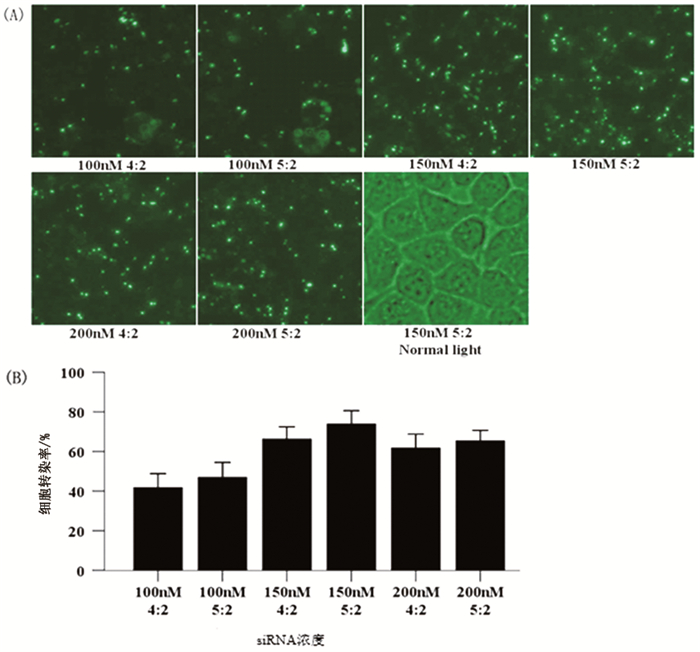
|
图 1 HeLa细胞中siRNA-NC转染率观察(400×) 注:(A)荧光显微镜观察;(B)流式细胞技术检测 |
siRNA并感染病毒36h后HeLa细胞的病变情况,发现三个对照组siRNA-NC、mock和virus only中大部分的HeLa细胞均已出现病变。siRNA-1、siRNA-2、siRNA-6三组细胞病变情况与阴性对照组相似,基本未表现出保护效果。siRNA-5组病变最少,siRNA-8和siRNA-4组病变次之(见图 2)。与倒置显微镜下观察的细胞病变结果相似,MTT法检测结果显示三个对照组mock、siRNA-NC、virus only和siRNA-1、siRNA-2、siRNA-6处理组中细胞死亡率高达80%,而其它组细胞的死亡率排序依次为:siRNA-11>siRNA-10>siRNA-9>siRNA-3>siRNA-4>siRNA-8>siRNA-5。因siRNA-1、siRNA-2、siRNA-6三组siRNA未表现出保护效果,故在此处淘汰此三组,后期不再进行检测和实验。所有数据进行了三个重复实验,三次重复之间进行比较,*P < 0.05,**P < 0.01。
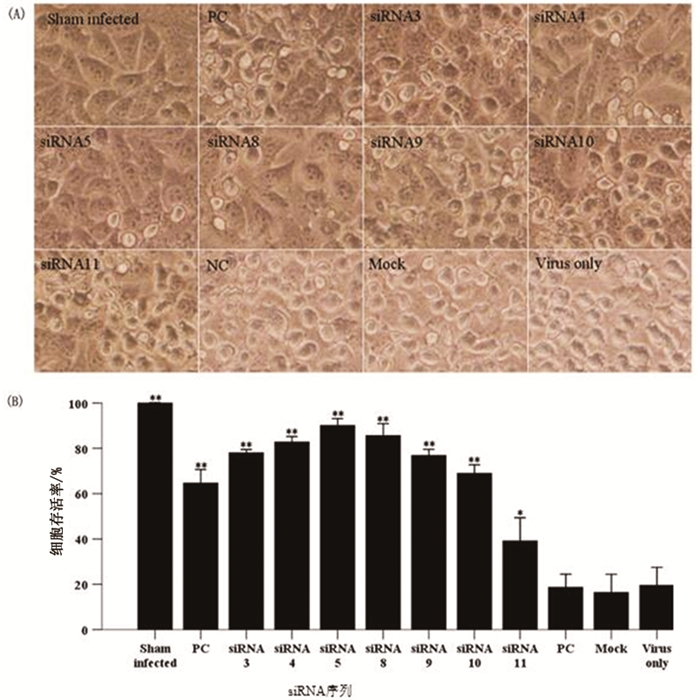
|
图 2 siRNA对CVB3导致的细胞病变的抑制 注:A, 倒置显微镜下观察HeLa的细胞病变情况(200×); B, MTT法检测不同组细胞存活率 |
Real-time PCR方法结果显示siRNA-5对病毒的抑制率最高,为84%,siRNA-8次之,为82%,siRNA-11最低仅为28%。siRNA-PC、siRNA-3、siRNA-4、、siRNA-9和siRNA-10分别为:62%、72%、80%、72%和63%。所有数据进行了三个重复实验,三次重复之间进行比较,*P < 0.05,**P < 0.01(图 3)。
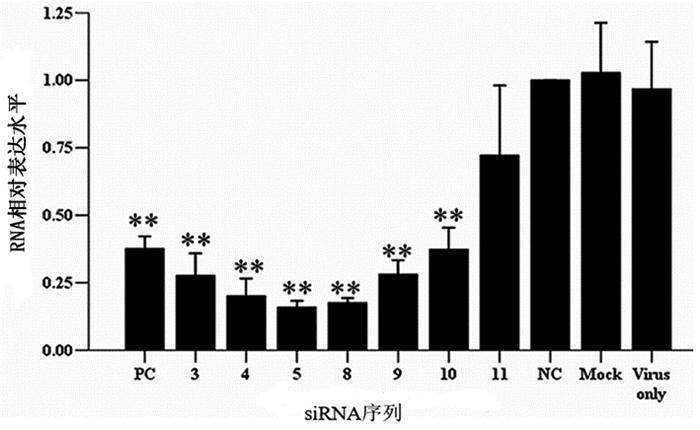
|
图 3 mRNA水平检测siRNA的抑制效果 |
VP1蛋白的特异性条带出现在所有感染CVB3的细胞组中,三个对照组virus only、mock和siRNA-NC处理的HeLa细胞中VP1蛋白表达的量相差不大,均较多。但siRNA-5、siRNA-8、siRNA-4处理的HeLa细胞中VP1蛋白的量明显减少。所有数据进行了三个重复实验,三次重复之间进行比较,*P < 0.05,**P < 0.01。见图 4。
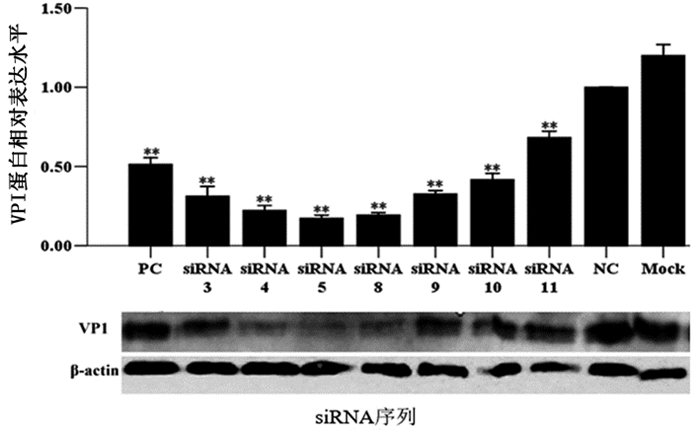
|
图 4 蛋白水平检测siRNAsiRNA对CVB3的抑制 |
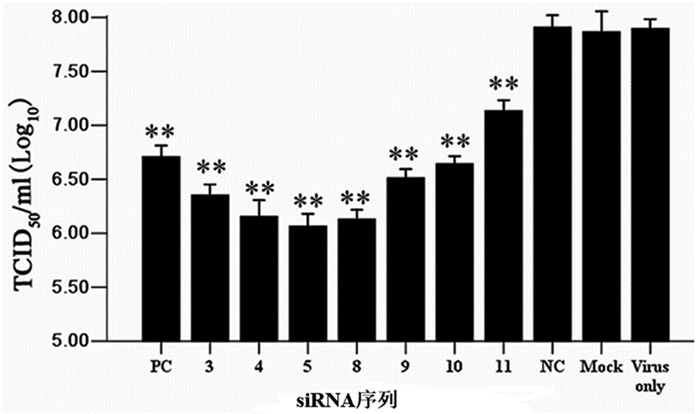
毒价滴定结果显示,siRNA-11对病毒的抑制率最低,与siRNA-NC相比,CVB3的毒价只降低了0.7log10。其中siRNA-5的抑制率最高,CVB3的毒价下降了2log10。与MTT、CPE、Western blot和real-time PCR结果相吻合,siRNA对CVB3的抑制率依是:siRNA-5>siRNA-8>siRNA-4>siRNA-3>siRNA-9>siRNA-10>siRNA-11(见图 5)。所有数据进行了三个重复实验,三次重复之间进行比较,*P < 0.05,**P < 0.01。
|
图 5 siRNA对CVB3毒价的抑制 |
柯萨奇病毒B3呈球形,直径约22~30nm,为单股正链RNA,全长由7396个碱基组成,具有mRNA活性和感染性[17]。依功能分区为5′端非编码区(5′-untranslated region,5′-UTR)、P1区、P2区、P3区、3′端非编码区(3′-UTR)和一个Poly(A)尾,CVB3可以引起很多疾病,至今尚无有效治疗手段。部分研究表明,在哺乳类细胞,甚至人类细胞中导入21~23个核苷酸(nucleotide, nt)的短双链RNA(small interfering RNA, siRNA)可使靶向基因的表达产生明显的特异性强抑制效应,而且不会诱发免疫反应和细胞毒性,因此siRNA应用于临床治疗成为可能[18]。
CRE对病毒复制是必不可缺的,在CVB3基因组中3’-UTR区、5’-UTR区和2C区各有一个CRE存在。研究发现一旦病毒的CRE遭到破坏,其在宿主体内的复制将会受到严重影响,因3’-UTR区和5’-UTR区不是设计siRNA的适宜靶点,故很多研究选取2C区的CRE为靶点进行研究。Merl等[11]的研究已证实在EV71和CAV24两种病毒中在其基因序列中的2C区CRE部分设计的siRNA序列有效的沉默了这两种病毒的致病性。然而针对CVB3目前仅有Kim等[12]组成的团队在其2C区的CRE部分发现了一条沉默效率较高的siRNA靶序列,那么在CVB3基因组中2C区CRE以外的部分是否是设计siRNA的有效靶点迄今为止没有任何研究证实。为此,本研究设计了siRNA-5和siRNA-6两条siRNA序列,它们均以2C区CRE以外的基因序列为靶点。本文结果显示本次筛选的11条siRNA序列中,siRNA-5对CVB3的抑制率最高,其沉默效率高达84%,经siRNA-5处理的HeLa细胞组其细胞活率达到了86%。首次证实验证了CVB3基因组中2C区CRE以外的部分也是设计siRNA的有效靶点。
部分研究发现针以2A区为靶点设计的siRNA序列对病毒的沉默效率较高[11]。本研究设计的siRNA-3以2A区为靶点,其对CVB3的沉默效率达到了72%。但本研究中以2C区为靶点设计的siRNA-5对CVB3的沉默效率远远高于在2A区设计的siRNA-3,因此,本研究团队推测CVB3基因组中2A区可能并非是设计siRNA靶序列最理想的区域。
本文发现了5条新的siRNA靶序列,它们对CVB3的沉默效率均较高,同时首次证实了2C区CRE以外的部分也是设计siRNA的理想靶点。本研究为防治CVB3感染引起的病毒性疾病提供了新的靶点和策略。
| [1] |
李红艳, 刘银芝, 王慧杰, 等. 羟基红花黄色素A对CVB3病毒性心肌炎小鼠血清TNF配体相关分子-1 A及IFN-γ表达的影响[J]. 世界中西医结合杂志, 2018, 13(10): 64-66. DOI:10.13935/j.cnki.sjzx.181016 |
| [2] |
Fung G, Luo H, Qiu Y, et al. Myocarditis[J]. Circu Res, 2016, 118(3): 496-514. DOI:10.1161/circresaha.115.306573 |
| [3] |
古宏标, 张丽蓉, 胡太平, 等. "先天性因素"不宜作为独立的致病因素提出[J]. 中国医院用药评价与分析, 2016(S1): 213. DOI:10.14009/j.issn.1672-2124.2016.s1.184 |
| [4] |
Luo X, Yao H, Song J, et al. Coxsackievirus B3 infection triggers autophagy through 3 pathways of endoplasmic reticulum stress[J]. Biomed Environ Sci, 2018, 31(12): 867-875. DOI:10.3967/bes2018.115 |
| [5] |
Ren L, Ding S, Song Y, et al. Profiling of rotavirus 3'UTR-binding proteins reveals the ATP synthase subunit ATP5B as a host factor that supports late-stage virus replication[J]. Biol Chem, 2019(online). DOI:10.1074/jbc.RA118.006004 |
| [6] |
王帅, 孔霞, 张国安, 等. RNAi介导ObR基因沉默对人乳头状甲状腺癌K1细胞增殖与侵袭能力的影响[J]. 济宁医学院学报, 2015, 38(2): 99-102. DOI:10.3969/j.issn.1000-9760.2015.02.006 |
| [7] |
秦燕霞, 李京, 柳新平. siRNA沉默B7-H4基因对人卵巢癌细胞增殖能力的影响[J]. 济宁医学院学报, 2016, 39(5): 320-323. DOI:10.3969/j.issn.1000-9760.2016.05.005 |
| [8] |
Ahn J, Jun ES, Lee HS, et al. A small interfering RNA targeting coxsackievirus B3 protects permissive HeLa cells from viral challenge[J]. Virol, 2005, 79(13): 8620-8624. DOI:10.1128/jvi.79.13.8620-8624.2005 |
| [9] |
Merl S, Michaelis C, Jaschke B, et al. Targeting 2A protease by RNA interference attenuates coxsackieviral cytopathogenicity and promotes survival in highly susceptible mice[J]. Circulation, 2005, 111(13): 1583-1592. DOI:10.1161/01.cir.0000160360.02040.ab |
| [10] |
Merl S, Wessely R. Anti-coxsackieviral efficacy of RNA interference is highly dependent on genomic target selection and emergence of escape mutants[J]. Oligonucleotides, 2007, 17(1): 44-53. DOI:10.1089/oli.2007.0057 |
| [11] |
Yuan J, Cheung PK, Zhang HM, et al. Inhibition of coxsackievirus B3 replication by small interfering RNAs requires perfect sequence match in the central region of the viral positive strand[J]. Virol, 2005, 79(4): 2151-2159. DOI:10.1128/jvi.79.4.2151-2159.2005 |
| [12] |
Kim YJ, Ahn J, Jeung SY, et al. Recombinant lentivirus-delivered short hairpin RNAs targeted to conserved coxsackievirus sequences protect against viral myocarditis and improve survival rate in an animal model[J]. Virus Genes, 2008, 36(1): 141-146. DOI:10.1007/s11262-007-0192-y |
| [13] |
Knowlton KU, Jeon ES, Berkley N, et al. Mutation in the puff region of VP2 attenuates the myocarditic phenotype of an infectious cDNA of the Woodruff variant of coxsackievirus B3[J]. Virol, 1996, 70(11): 7811-7818. DOI:10.1111/j.1365-2893.1996.tb00107.x |
| [14] |
Slifka MK, Pagarigan R, Mena I, et al. Using recombinant coxsackievirus B3 to evaluate the induction and protective efficacy of CD8+ T cells during picornavirus infection[J]. Virol, 2001, 75(5): 2377-2387. DOI:10.1128/jvi.75.5.2377-2387.2001 |
| [15] |
Elbashir SM, Lendeckel W, Tuschl T. RNA interfering is mediated by 21-and-22-nucleotide RNAs[J]. Genes Dev, 2001, 15(2): 188-200. DOI:10.1101/gad.862301 |
| [16] |
Reynolds A, Leak D, Boese Q, et al. Rational siRNA design for RNA interference[J]. Nat Biotechnol, 2004, 22(3): 326-330. DOI:10.1038/nbt936 |
| [17] |
Kim K, Kanno T, Chapman NM, et al. Genetic determinants of virulence in the group B coxsackieviruses[J]. Future Virol, 2006, 1(15): 597-604. DOI:10.2217/17460794.1.5.597 |
| [18] |
Grimm D. Small silencing RNAs:state-of-the-art[J]. Adv Drug Deliv Rev, 2009, 61(9): 672-703. DOI:10.1016/j.addr.2009.05.002 |

