海洋微生物种类丰富,参与海洋生态循环,与海洋环境有着密切联系,对维护海洋生态系统的稳定起重要作用[1]。近岸海域由于其有机物含量较高,为微生物的生长繁殖提供了充足的营养物质,具有广阔的开发利用价值。Williams等[2]从近岸海域分离得到的蓝细菌和放线菌所分泌的大量化合物具有治疗肿瘤和传染病的作用,可作为宝贵的药物来源;信艳娟等[3]从大连湾原油污染海域分离得到高效的石油烃降解菌,可用于海洋石油污染的降解,具有良好的应用前景。因此,研究近岸海域的微生物群落,能更全面地了解相关海域的微生物分布情况和生态意义,有助于发现和利用微生物资源。
目前,针对日照近岸海域的微生物群落结构研究较少。本文通过构建16S rRNA基因克隆文库的方法对该海域微生物的群落结构进行解析,以了解和认识该海域微生物的主要分类及作用,为合理开发利用海洋微生物资源提供参考。
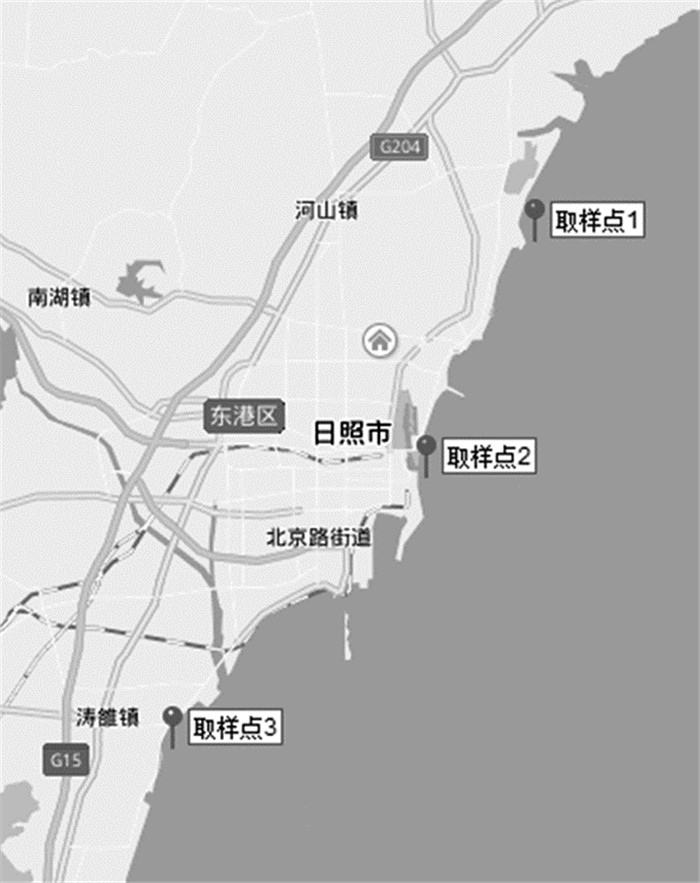
1 材料和方法 1.1 材料实验所用海水取自日照近岸海域3个取样点(图 1),基本沿海岸线均匀分布,每个取样点周边取样3次。
|
图 1 取样点分布示意图 |
培养基:LB培养基,SOB培养基,SOC培养基(在SOB培养基中添加10ml灭菌的1mol/L葡萄糖)。
主要试剂:水样DNA提取试剂盒(Mo Bio Laboratories,Inc.,USA),TAE缓冲液(北京索莱宝科技有限公司),大肠杆菌DH-5α感受态细胞(北京索莱宝科技有限公司),X-Gal(北京索莱宝科技有限公司),IPTG(北京索莱宝科技有限公司),凝胶回收试剂盒(宝生物工程有限公司),TA克隆载体pMD-19T(宝生物工程有限公司),限制性内切酶MspⅠ(宝生物工程有限公司)。
主要仪器:PCR仪(美国伯乐公司),冷冻高速离心机(美国热电公司),恒温振荡培养箱(南通海伦生物器材制造有限公司),超净工作台(北京华威兴业科技有限公司),凝胶成像仪(美国伯乐公司),核酸电泳仪(美国伯乐公司)。
1.2 方法 1.2.1 海水总DNA提取将海水样品进行等体积混合均匀后,用一次性灭菌的注射器将100ml样品用0.22μm滤膜过滤,将其中的微生物截留到滤膜表面及滤孔中。用镊子取下滤膜,参照水样DNA提取试剂盒的操作步骤提取样品中的总DNA。提取的总DNA用1%琼脂糖凝胶、100V电压进行电泳检测。
1.2.2 海水DNA扩增及产物纯化以提取的总DNA为模板,采用细菌16S通用引物27F(AGRGTTTGATCMTGGCTCAG)和1387R(GGGCGGWGTGTACAAGGC)进行扩增(反应体系见表 1)。反应条件为:95 ℃预变性3min,95 ℃变性30s,56 ℃退火30s,72 ℃延伸1.5min,循环28次,然后72 ℃终延伸7min。扩增结束后,产物用1%琼脂糖凝胶、100V电压进行电泳检测。
| 表 1 海水DNA扩增反应体系 |
为了防止扩增过程中产生的非特异性扩增而影响后续的实验,需对目的条带进行回收、纯化,参照凝胶回收试剂盒的操作步骤进行。纯化后的产物用1%琼脂糖凝胶、100V电压进行电泳检测,以验证扩增产物是否纯化成功。
1.2.3 阳性克隆筛选将扩增产物与克隆载体pMD19-T连接,体系为pMD19-T Vector 1μl,PCR产物4μl,Solution Ⅰ溶液5μl,4 ℃连接过夜。将过夜连接的产物加入到100μl大肠杆菌感受态细胞中,冰上放置30min。从SOC培养基中取890μl加入到激活的感受态细胞中,37 ℃、180 rpm摇床培育1h。分别取50μl、100μl、300μl、500μl培养液涂布于含有8μl IPTG、40μl X-Gal、含0.1%氨苄的LB平板上,37 ℃培养,培养后的平板放入4 ℃冰箱中显色1h,从长满克隆的平板上随机挑选64个白色克隆划线至含0.1%氨苄的平板上,37 ℃培养至菌落长出。每个克隆分别进行菌落DNA扩增(反应体系见表 2),所用载体引物为M13-47(CGCCAGGGTTTTCCCAGTCACGAC)和RV-M(AGCGGATAACAATTTCACACAGGA),反应条件为:95 ℃预变性3min,95 ℃变性30s,56 ℃退火30s,72 ℃延伸1.5min,循环28次,72 ℃终延伸7min。扩增产物用1%琼脂糖凝胶、100V电压进行电泳检测假阳性克隆。
| 表 2 菌落DNA扩增反应体系 |
菌落DNA模板制作的方法为:挑克隆到10μl无菌水中,95 ℃水浴10min,冰上放置3min,5000rpm离心2min,取上清即为模板。
1.2.4 酶切分型用限制性内切酶Msp I对扩增产物进行酶切,体系为:10×Buffer 0.8ml,MspⅠ 0.4μl,扩增产物3.6μl,补无菌水至8μl,37 ℃消化16h后,用3%琼脂糖凝胶、65V电泳约1h后进行检测。酶切后产物的电泳图谱呈现多态性,对电泳图谱进行归类分析,图谱一致视为同种克隆。
1.2.5 目的基因测序分别从同种克隆里选出一个接种于含0.1%氨苄的LB液体培养基中,37 ℃、200rpm摇床中培养8h后,取2ml菌液,送至苏州金唯智生物科技有限公司测序。
1.2.6 测序结果分析将所测得的序列提交至NCBI(https://blast.ncbi.nlm.nih.gov/)进行比对,根据比对结果分析微生物的群落结构。
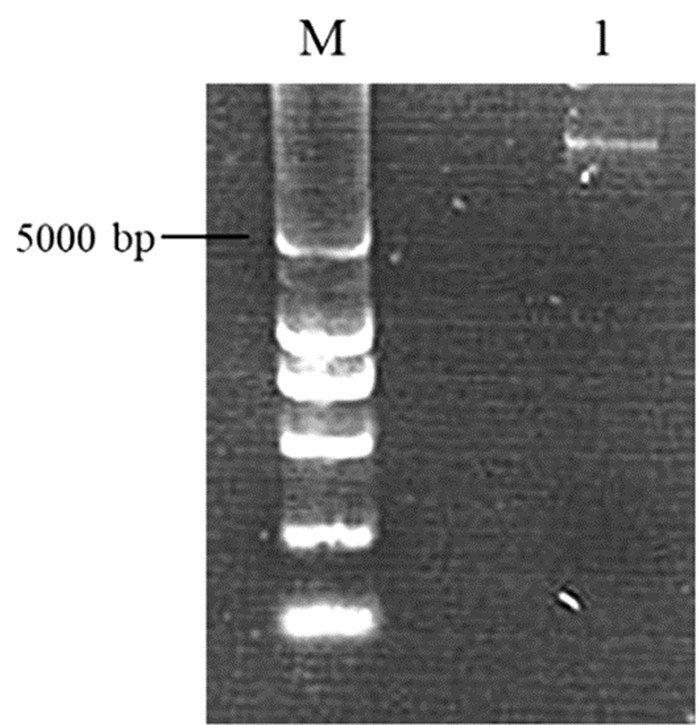
2 结果与讨论 2.1 海水总DNA提取海水样品中的总DNA提取结果如图 2所示,编号为1的条带为提取的总DNA,可见明显条带,能够满足后续分析需要。
|
图 2 海水样品总DNA |
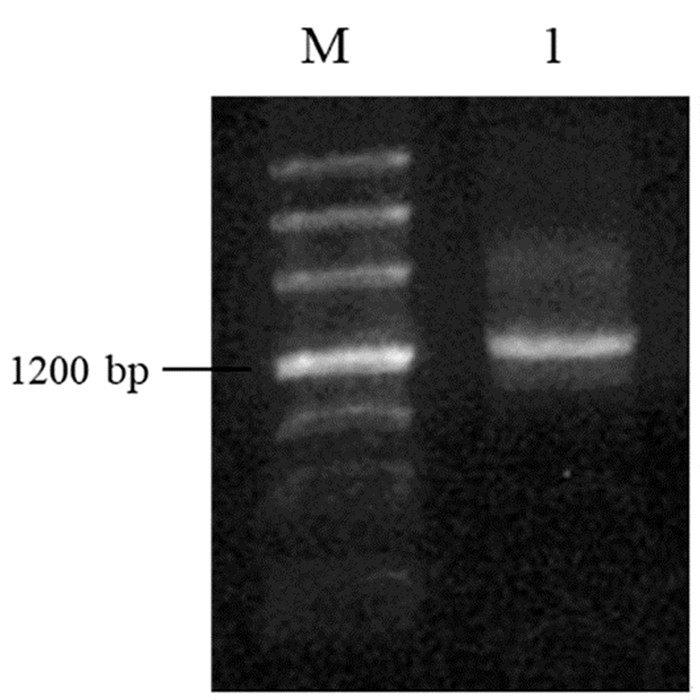
扩增产物如图 3中条带1所示,可见条带较亮,但存在轻微拖尾现象。
|
图 3 海水DNA扩增产物 |
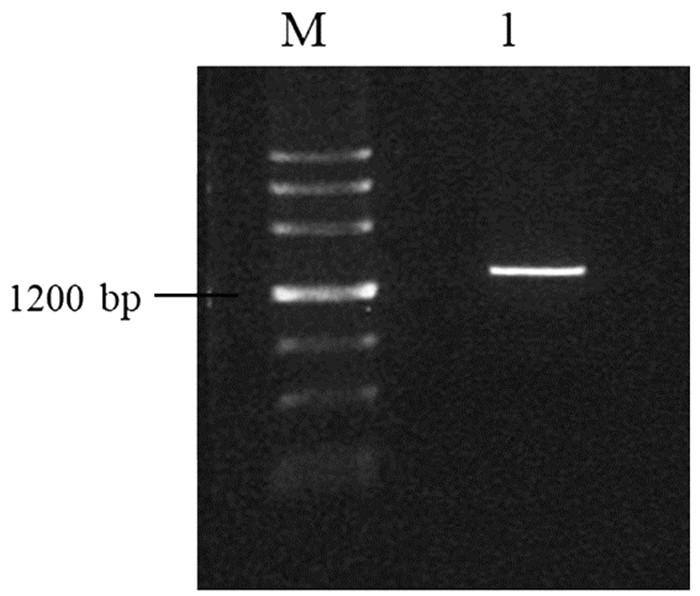
对目的条带进行回收、纯化后结果如图 4条带1所示。可见单一条带,无拖尾现象,纯化效果好。
|
图 4 纯化后的扩增产物 |
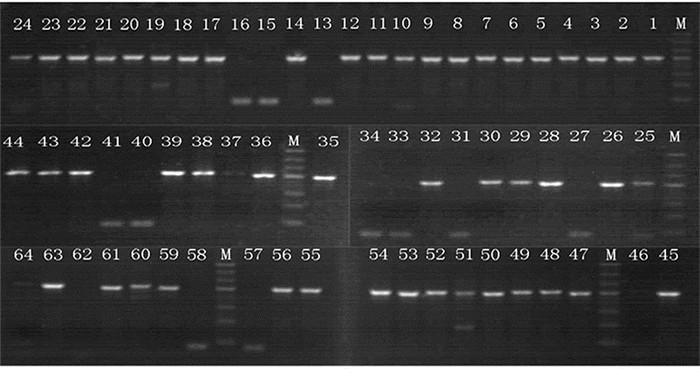
由于T载体自连等原因,往往白色克隆中会有部分是假阳性克隆。如图 5所示,共有15个假阳性克隆(克隆编号分别为:13、15、16、27、31、33、34、37、40、41、46、57、58、62、64)。
|
图 5 假阳性克隆电泳检测 |
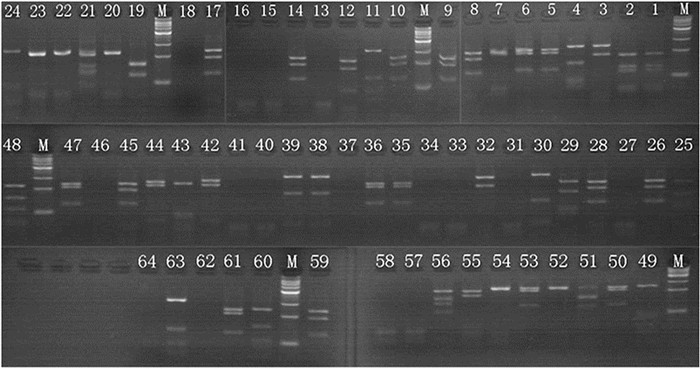
如图 6所示,酶切后产物的电泳图谱呈现多态性,将酶切带型一致的克隆归为一类,定义为一个分类单元,共分为15类,从每一类中任选1个克隆进行测序(测序的克隆编号:2、3、4、12、18、19、20、24、28、30、36、39、42、48、56)。
|
图 6 限制性内切酶MspⅠ酶切图谱 |
将所测得的序列提交至NCBI进行比对,比对结果见表 3。
| 表 3 测序比对结果 |
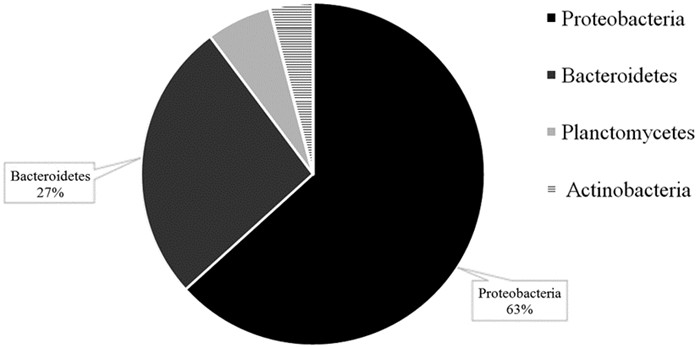
对测序结果进行统计分析,如图 7所示,日照近岸海域微生物群落组成来自4个门类。其中,变形菌门(Proteobacteria)所占比例最高,达63%;拟杆菌门(Bacteroidetes)次之,达27%;浮霉菌门(Planctomycetes)和放线菌门(Actinobacteria)所占比例较低,分别为6%和4%。
|
图 7 门水平群落组成 |
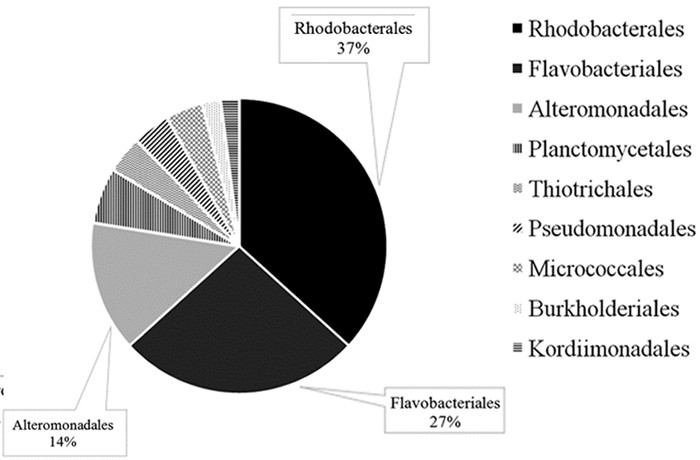
如图 8所示,日照近岸海域微生物群落组成来自9个目类。其中,红细菌目(Rhodobacterales)、黄杆菌目(Flavobacteriales)、交替单胞菌目(Alteromonadales)所占比例较高,分别达到37%、27%和14%;其余6个目类所占比例较低,均低于10%。
|
图 8 目水平群落组成 |
如图 9所示,日照近岸海域微生物群落组成来自十几个不同属类。其中,亚硫酸杆菌属(Sulfitobacter)所占比例最高,高达35%。海洋微生物作为生物活性物质的来源越来越受到国内外研究学者的重视[4]。龙聪等[5]对44株来源于中国东海的亚硫酸杆菌进行生物学活性分析。结果显示,其表现出显著的细胞毒活性及抗氧化活性,具有潜在的药用价值。Brakstad等[6]发现亚硫酸杆菌属的细菌与海洋原油的生物降解密切相关。高慧[7]选取全国13个不同油污染的地区水样研究发现,以柴油为唯一碳源,筛选出具有柴油降解能力的亚硫酸杆菌。另外,假交替单胞菌属(Pseudoalteromonas)和黄杆菌属(Flavobacterium)所占比例也较高,分别达到15%和12%。假交替单胞菌能分泌多种胞外活性物质,这些活性物质在防治有害藻类水华、生物防污、工业生产和开发抗菌和抗肿瘤海洋药物等方面展示了诱人的应用前景[8]。孙雪莹[9]从300株细菌中筛选出一株既能够产酶又拮抗灿烂弧菌的假交替单胞菌,且不产生溶血素,不具有潜在的致病性,可作为贝类生长的益生菌。姜肸等[10]从南海10个采样点筛选出6株对石油烃有较强降解能力的细菌,其中有两株为假交替单胞菌。许晓毅等[11]从长江近岸表层沉积物中分离出2株能以菲和荧蒽为碳源和能源生长的菌株,其中一株为黄杆菌属,且表现出非常强的多环芳烃降解能力。

|
图 9 属水平群落组成 |
通过构建16S rRNA基因克隆文库对日照近岸海域的微生物群落结构进行分析,结果显示此海域微生物多样性水平较高,主要包括亚硫酸杆菌属、假交替单胞菌属和黄杆菌属等。其中,亚硫酸菌属所占的比重最大,具有潜在的生态意义和药用价值。另外,能够降解石油烃的微生物比重较大,说明日照近岸海域的石油烃污染可能性较大,究其原因可能是日照作为新兴的港口城市,难免存在港口和船舶操作性含油污水的排放及油类作业泄露。本研究可以更好地了解和认识该海域微生物的主要分类及作用,并以此为依据分析日照近岸海域特征及海洋状况,达到合理开发利用海洋资源的目的,促进半岛蓝色经济区的可持续发展。
| [1] | Arrigo KR. Erratum:Marine microorganisms and global nutrient cycles[J]. Nature, 2005, 437(7057): 349. DOI:10.1038/nature04159 |
| [2] | Williams PG. Panning for chemical gold:marine bacteria as a source of new therapeutics[J]. Trends in Biotechnology, 2009, 27(1): 45–52. DOI:10.1016/j.tibtech.2008.10.005 |
| [3] | 信艳娟, 刘亚男, 吴佩春, 等. 一株原油降解菌的分离鉴定及降解特性研究[J]. 微生物学通报, 2013, 40(4): 558–566. |
| [4] | Romano G, Costantini M, Sansone C, et al. Marine microorganisms as a promising and sustainable source of bioactive molecules[J]. Marine Environmental Research, 2016, 128: 58. DOI:10.1016/j.marenvres.2016.05.002 |
| [5] | 龙聪, 卢小玲, 刘军华, 等. 中国东海亚硫酸杆菌属微生物的鉴定及生物学活性分析[J]. 第二军医大学学报, 2009, 30(10): 1106–1109. |
| [6] | Brakstad OG, Ldeng AG. Microbial diversity during biodegradation of crude oil in seawater from the North Sea[J]. Microbial Ecology, 2005, 49(1): 94–103. DOI:10.1007/s00248-003-0225-6 |
| [7] | 高慧. 海洋石油降解菌的多样性研究[D]. 济南: 山东大学, 2016. |
| [8] | 席宇, 朱大恒, 刘红涛, 等. 假交替单胞菌及其胞外生物活性物质研究进展[J]. 微生物学通报, 2005, 32(3): 108–112. |
| [9] | 孙雪莹. 虾夷扇贝幼体及育苗池水体细菌群落动态及潜在益生菌筛选[D]. 大连: 大连海洋大学, 2016. |
| [10] | 姜肸, 高伟, 李倩, 等. 南海高效石油降解菌的筛选及降解特性研究[J]. 环境科学学报, 2012, 32(7): 1572–1578. |
| [11] | 许晓毅, 苏攀, 姬宇, 等. 沉积物中2株多环芳烃降解菌的分离鉴定及其对菲、荧蒽的降解特性[J]. 环境工程学报, 2015, 9(3): 1513–1520. DOI:10.12030/j.cjee.20150386 |



