脑外伤的评价和治疗一直是法医学和临床医学工作者关注的科学问题。近年来,脑外伤的发生率逐年上升,对于脑外伤所引起的神经功能继发性损害逐渐得到关注[1]。随着“精准医学”概念的提出,脑外伤后法医学的“精准鉴定”以及临床治疗中的准确判断和治疗成为关键点。科学技术的不断发展和完善,使得蛋白质组学等高通量研究手段的应用范围不断扩大,这也为脑外伤后神经功能损害机制的研究提供了新的途径[2]。本文拟通过建立大鼠脑外伤动物模型,观察脑外伤后损伤组织的蛋白组学变化情况,分析外伤后神经功能异常的可能机制,并为脑外伤后的蛋白生物标志物的筛选提供基础数据。
1 材料和方法 1.1 动物及主要仪器8周龄雄性SD大鼠,体重300~350g,购于上海斯莱克实验动物公司。复合型静电轨道阱回旋共振液质联用仪(Orbitrap Elite LC-MS/MS, Thermo,美国); 冷凝浓缩仪(Labconco,美国); 冷冻离心机(CL-21R,Thermo,美国)。
1.2 主要试剂质谱测序级胰酶(美国Sigma公司); 氯乙酰胺(美国Sigma公司); TMT定量标记试剂盒(美国Thermo公司)。实验所用溶剂以及流动相均为LCMS级。
1.3 方法 1.3.1 动物模型制备及样本采集将每一次生物学重复的25只SD大鼠随机分成5组,包括损伤后1h、6h、24h、3d组以及对照组,每组5只。称重后10%水合氯醛(0.35ml/100g)腹腔注射麻醉。立体定位仪固定并用颅骨钻钻孔,通过自由落体打击器建立脑外伤模型。伤后1h、6h、24h、3d断头取脑,冰上分离伤侧皮质,-80℃冻存备用。
1.3.2 蛋白提取以及胰酶消化RIPA裂解液加入蛋白酶抑制剂以及磷酸酶抑制剂后,每样本加入200μl裂解液冰上裂解30min涡旋破碎组织后,12000g,4℃离心15min取上清液。BCA法测定蛋白浓度,用裂解液将样本浓度调整为1μg/μl。每个时间点的5个样本各取15μl混合,加入4倍体积丙酮沉淀过夜。离心后用8M尿素将沉淀溶解,进行还原烷基化反应以打开蛋白间的二硫键。按质量比50:1比例加入1mg/ml胰酶消化过夜。
1.3.3 肽段的标记与除盐对消化后的肽段进行TMT同位素标记,对照组用TMT-126进行标记(5只大鼠样本混合后分成4组与损伤组配对),损伤组用TMT-127进行标记(1h、6h、24h、3d),室温避光反应2~3h。将标记后的肽段通过除盐柱,除去肽段溶液中的盐分,对肽段浓度定量后转入进样瓶冷凝干燥,-20℃保存待质谱检测。
1.3.4 酶解后的肽段样品质谱检测根据肽段浓度的定量结果溶于0.1%甲酸中,流动相A是双蒸水稀释的0.1%甲酸,流动相B是80%乙腈和0.1%甲酸,上样量为6μl,液相分离条件为:0~10min,6%B相;10~130min,6%~44%B相;130~140min,98%B相;140~150min,6%B相。Xcalibur在正电荷模式下自动采集数据一级的MS谱图,设置母离子扫描分辨率为120000,采用HCD碎裂方式对强度最高的15个离子进行二级质谱分析。通过对碎片信息的采集,得到多肽的二级谱图,进行数据库搜库获得蛋白信息,肽段匹配的假阳性率(FRD < 1%),共进行3次生物学重复,对于每个时间点,选取3次均鉴定到的蛋白作为最终结果,以确保实验数据的准确性。
1.3.5 生物信息学分析与富集通过DAVID在线数据库,对每个时间点所鉴定到的差异蛋白(损伤组和对照组相比蛋白表达上调1.2倍或下调0.8倍)进行生物学过程、细胞构成、分子功能以及代谢通路的富集,分析外伤后的蛋白变化差异。
2 结果 2.1 脑外伤后蛋白表达变化脑外伤发生后蛋白表达出现了变化,筛选出每个时间点中上调1.2倍或下调0.8倍的蛋白(P < 0.05),且所进行的3次生物学重复均涵盖的蛋白作为差异性蛋白。结果显示:脑外伤发生后在1h、6h、24h、3d分别有83、66、127、155个蛋白的表达发生差异性表达。见图 1。

|
图 1 损伤后1h、6h、24h、3d差异蛋白数量 |
根据所挑选出的差异蛋白,运用DAVID在线数据库进行分析,对差异蛋白进行GO富集,根据生物过程、细胞构成、分子功能对差异蛋白进行分类。在分子功能方面,差异蛋白主要发挥了结合、分子结构的激活、接触反应的激活等作用。在生物学过程中,大量差异表达蛋白与细胞过程、代谢过程、细胞构成调节、生物合成有关。在细胞构成方面差异蛋白来源于细胞膜、大分子复合体、细胞器等结构。见图 2。

|
图 2 脑外伤后1h、6h、24h、3d的GO富集分析 |
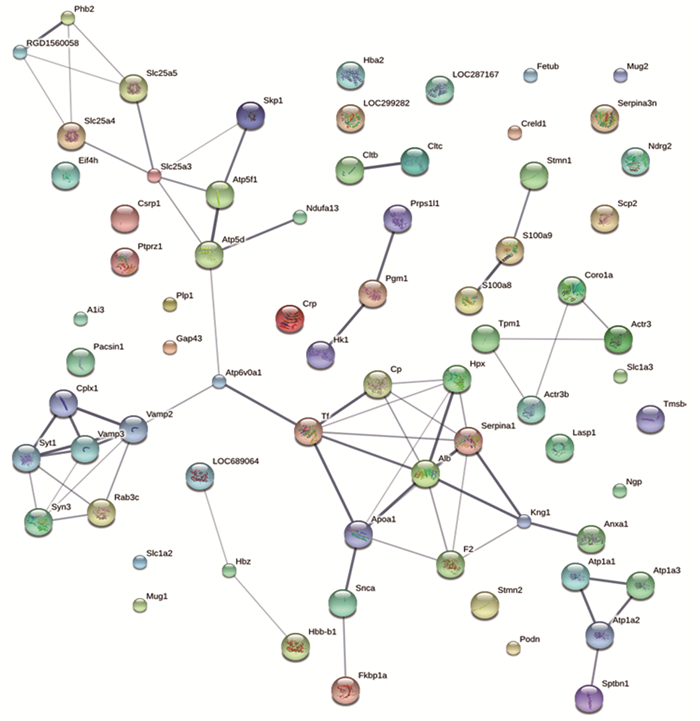
根据实验中不同时间点差异蛋白的数量情况,我们发现在24h以及3d时,差异蛋白的数量显著增加。因此,进一步分析差异蛋白的相互作用关系。从分析结果可以看出,在脑外伤后24h,血清白蛋白(Alb)、囊泡结合膜蛋白3(Vamp3) 可以作为蛋白相互作用以及影响的中心物质;在外伤后3d,肌动蛋白2(Actbl2)、微管蛋白(Tuba1b)等处于相互作用的中心。见图 3、图 4。
|
图 3 脑外伤后24h差异蛋白相互作用示意图 |

|
图 4 脑外伤后3d差异蛋白相互作用示意图 |
随着蛋白组学、代谢组学等高通量研究手段相继被引入到医学研究当中,实现了医学与生物学的交互和融合,有利于从分子水平阐述疾病和损伤的发生发展过程。高通量所产生的大数据结合生物信息学的分析可以更快速全面的分析研究疾病产生的机制。通过蛋白组学的策略和方法,研究脑外伤后的蛋白变化水平,可以更全面、更完整的了解脑外伤发生后蛋白代谢的情况,得到全面的物质变化信息[3];同时运用生物信息学的分析方法,可以更直观地看出蛋白调控差异。
本文通过定量蛋白组学的研究方法,对损伤组织以及非损伤的对照组织进行了相对定量比较。根据TMT-126与TMT-127的分子量相同,但质荷比不同的特性,因而可以通过肽段二级碎裂谱图进行相对定量,从而反映出不同损伤时间点的蛋白表达情况。
本实验的生物信息学分析结果显示,各个时间点中,差异蛋白参与了多方面的调控。从差异蛋白的分子构成上来看,较多的蛋白来源于细胞器。细胞发生损伤后,可以通过囊泡等途径进行降解,细胞器破裂[4], 蛋白释放。因此,在实验的分析结果中可以明确地看出差异蛋白的来源与分布,为今后外伤后亚细胞结构的研究以及功能性蛋白在亚细胞结构上的定位提供基础。同时从生物学过程以及分子功能方面来看,参与结合功能的蛋白以及代谢相关途径所富集到的差异蛋白较为明显,说明外伤后,脂类代谢,能量合成与供应等生物学过程发生显著变化,并可能成为脑外伤后继发性神经损害的机制之一[5-6]。差异蛋白在结合方面的分子功能包括离子结合、氧结合等,这类结合更多地表现为非共价结合,通过这种结合方式可以调节体内代谢过程[7]。
对与差异蛋白的相互作用,在24h和3d中,Vamp3以及Actbl2等与多种差异蛋白可以发生相互作用。对于膜突触结合蛋白1(Syt1) 来说,当与钙离子结合后,可同时促进神经传递,并使质膜以及1-SNARE形成复合体产生分子功能[8]。通过对脑外伤后所表达的差异蛋白进行互作分析,可以进一步深入分析代谢通路间的作用联系[9-10],同时可能成为损伤后标志物研究的新途径。
司法鉴定与临床治疗虽为两个方向,却是相互补充、不可分离的两个整体。不同的损伤程度、甚至损伤引起的死亡,其相关的标志物可能成为鉴定推断的可靠依据,而对相关标志物的调节,可能成为临床治疗的新靶点、新依据[11-12]。本阶段实验通过质谱、生物信息学等高通量方式对脑外伤后的物质变化进行初步研究,为今后脑外伤生物标记物的研究提供基础数据,同时也有助于对外伤后的蛋白相互作用的确切机制进行更深入的发掘。
| [1] | Grauwneijer E, Heijenbrok-kal M, Haitsma I, et al. Employment outcome ten years after moderate to severe traumatic brain injury:a prospective cohort study[J]. J Neurotrauma, 2017. DOI:10.1089/neu.2016.4846 |
| [2] | Sarkis GA, Mangaonkar MD, Moghieb A, et al. The application of proteomics to traumatic brain and spinal cord injuries[J]. Curr Neurol Neurosci Rep, 2017, 17(3): 23. DOI:10.1007/s11910-017-0736-z |
| [3] | Xiong XG, Liang Q, Zhang C, et al. Serum proteome alterations in patients with cognitive impairment after traumatic brain injury revealed by iTRAQ-based quantitative proteomics[J]. Biomed Res Int, 2017, 2017: 8572509. DOI:10.1155/2017/8572509 |
| [4] | Levine J, Kwon E, Paez P, et al. Traumatically injured astrocytes release a proteomic signature modulated by STAT3-dependent cell survival[J]. Glia, 2016, 64(5): 668–694. DOI:10.1002/glia.22953 |
| [5] | Hill R L, Singh I N, Wang J A, et al. Time courses of post-injury mitochondrial oxidative damage and respiratory dysfunction and neuronal cytoskeletal degradation in a rat model of focal traumatic brain injury[J]. Neurochemistry International, 2017. DOI:10.1016/j.neuint.2017.03.015 |
| [6] | Roux A, Muller L, Jackson SN, et al. Mass spectrometry imaging of rat brain lipid profile changes over time following traumatic brain injury[J]. J Neurosci Methods, 2016, 272: 19–32. DOI:10.1016/j.jneumeth.2016.02.004 |
| [7] | Kidd RD, Russell JE, Watmough NJ, et al. The role of beta chains in the control of the hemoglobin oxygen binding function:chimeric human/mouse proteins, structure, and function[J]. Biochemistry, 2001, 40(51): 15669–15675. DOI:10.1021/bi011329f |
| [8] | Choi UB, Strop P, Vrljic M, et al. Single-molecule FRET-derived model of the synaptotagmin 1-SNARE fusion complex[J]. Nat Struct Mol Biol, 2010, 17(3): 318–324. DOI:10.1038/nsmb.1763 |
| [9] | Foster L J, Rudich A, Talior I, et al. Insulin-dependent interactions of proteins with GLUT4 revealed through stable isotope labeling by amino acids in cell culture (SILAC)[J]. Journal of Proteome Research, 2006, 5(1): 64–75. DOI:10.1021/pr0502626 |
| [10] | Ventadour S, Jarzaguet M, Wing SS, et al. A new method of purification of proteasome substrates reveals polyubiquitination of 20 S proteasome subunits[J]. J Biol Chem, 2007, 282(8): 5302–5309. DOI:10.1074/jbc.M610005200 |
| [11] | Westwood Sarah, Liu Benjamine, Baird Alison L, et al. The influence of insulin resistance on cerebrospinal fluid and plasma biomarkers of Alzheimer's pathology[J]. Alz Res Therapy, 2017, 9(1). DOI:10.1186/s13195-017-0258-6 |
| [12] | Byrum SD, Burdine MS, Orr L, et al. Time-and radiation-dose dependent changes in the plasma proteome after total body irradiation of non-human primates:Implications for biomarker selection[J]. PLoS One, 2017, 12(3): e0174771. DOI:10.1371/journal.pone.0174771 |




 陶陆阳, 男, 教授, 博士研究生导师。苏州大学医学部法医学系主任, 学科带头人。哈佛大学医学院麻省总医院神经科学中心访问学者, 哈佛大学麻省总医院脑外伤实验室博士后。苏州大学“衰老与神经疾病实验室”学术方向带头人。从事法医病理学创伤细胞/分子生物学以及创伤行为学电生理研究。担任《法医学杂志》编委、Neuronal Regeneration Research(SCI收录)执行编委、Research and Reports in Forensic Medical Science编委、《苏州大学学报(医学版)》编委。参编卫生部“十一五”、“十二五”规划教材《法医学》、《刑事科学技术》等人民卫生出版社著作5部。近年来在Srtoke、JCBFM、Neuroscience、J Forensic Science等发表论文60余篇, 在法医学领域和神经科学领域享有一定的威望
陶陆阳, 男, 教授, 博士研究生导师。苏州大学医学部法医学系主任, 学科带头人。哈佛大学医学院麻省总医院神经科学中心访问学者, 哈佛大学麻省总医院脑外伤实验室博士后。苏州大学“衰老与神经疾病实验室”学术方向带头人。从事法医病理学创伤细胞/分子生物学以及创伤行为学电生理研究。担任《法医学杂志》编委、Neuronal Regeneration Research(SCI收录)执行编委、Research and Reports in Forensic Medical Science编委、《苏州大学学报(医学版)》编委。参编卫生部“十一五”、“十二五”规划教材《法医学》、《刑事科学技术》等人民卫生出版社著作5部。近年来在Srtoke、JCBFM、Neuroscience、J Forensic Science等发表论文60余篇, 在法医学领域和神经科学领域享有一定的威望